Publicaciones SERIDA
Microorganismos de origen sidrero, recursos genéticos microbianos, al servicio de la biotecnología (II)
PANDO BEDRIÑANA, R.; VALDERAS HERRERO, M.T.; SUAREZ VALLES, B.
La mayoría de las propiedades tecnológicas que poseen los microorganismos utilizados en la industria agroalimentaria son cepa-dependiente. Este hecho evidencia la importancia de preservar y mantener las colecciones de cultivo, apoyando la búsqueda de nuevos aislados y los estudios de conservación de microorganismos.
El valor de las Colecciones de Cultivo Microbianas aumenta y se enriquece conforme se realizan actividades de investigación destinadas a la identificación y caracterización de los microorganismos que las constituyen. En este sentido, desde los años 2000, el Área de Tecnología de los Alimentos viene realizando, de forma paulatina, la identificación y caracterización de aislados de origen sidrero que constituyen la Colección de Cultivos Autóctonos del SERIDA.
Identificación de recursos microbianos
En la actualidad, la identificación de microorganismos se realiza mediante técnicas de biología molecular. Estos métodos analizan el genoma de los microorganismos de manera independiente al estado fisiológico de las células. Tienen como principal ventaja producir menos ambigüedades e incorrecciones que los métodos convencionales de identificación, en los que las características analizadas están fuertemente influenciadas por las condiciones de cultivo.
Las dos técnicas utilizadas para identificar a nivel de género o especie los recursos de origen sidrero son: el análisis de restricción de la región ribosómica 5,8S (levaduras) y la secuenciación de la región ribosómica 16S (bacterias lácticas).
En el caso de las levaduras la metodología consiste en amplificar mediante PCR la región 5,8S-ITS del ADN ribosomal y luego digerir dicha región con enzimas de restricción (Figura 1). Las diferencias de tamaño de las secuencias nucleotídicas, visualizadas mediante electroforesis, dan lugar a patrones especie-específicos (Esteve-Zarzoso y col., 1999). Finalmente la identificación se obtiene comparando los tamaños moleculares de los fragmentos de ADN con cepas de referencia y con la base de datos Yeast-id (http://www.yeast-id.com). Con esta técnica se han identificado en torno a 2000 levaduras detectándose la presencia, en sidras asturianas, de 14 especies (Tabla 1).
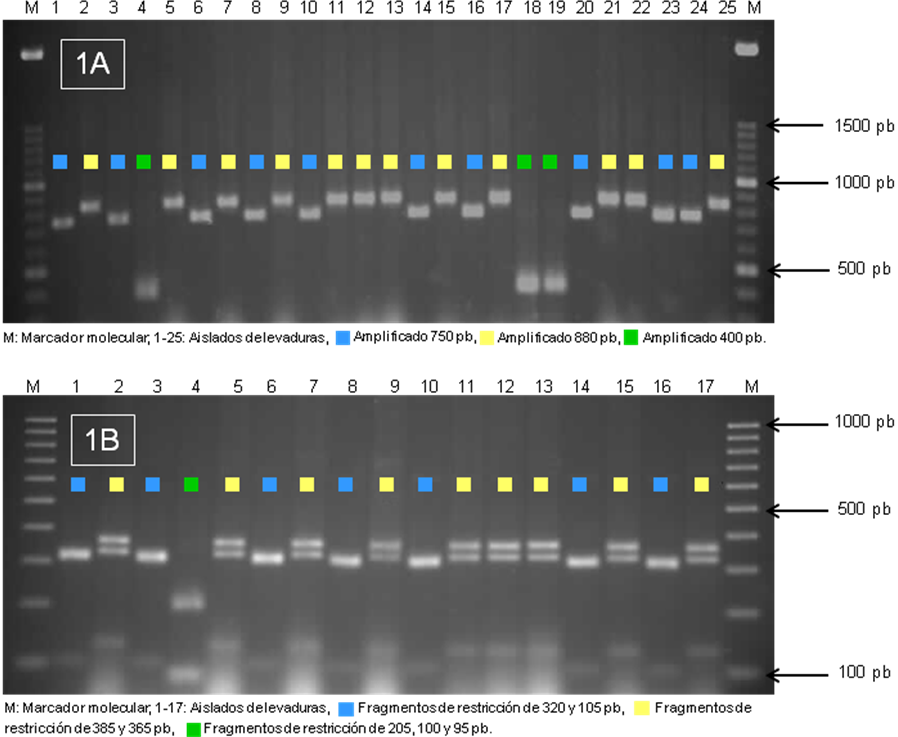
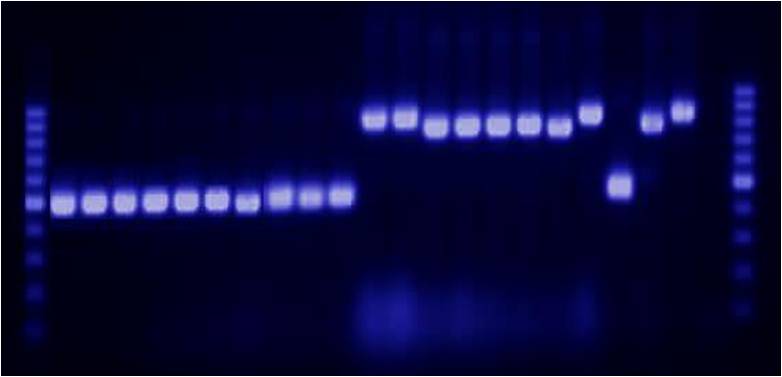
Figura 1: Perfiles de amplificación y restricción de la región ribosómica 5,8S-ITS de levaduras visualizados mediante electroforesis. 1A: Amplificados obtenidos en la reacción de PCR. 1B: Fragmentos de restricción derivados de la actividad del enzima Cfo I sobre los amplificados.
Tabla 1: Especies de levaduras y bacterias lácticas a las que pertenecen los recursos microbianos de origen sidrero.
| Levaduras | Bacterias lácticas |
| Candida glaebosa | Lactobacillus collinoides |
| Candida parapsilosis | Lactobacillus diolivorans |
| Candida vini | Lactobacillus hilgardii |
| Dekkera bruxellensis |
Lactobacillus plantarum/pentosus |
| Hanseniaspora osmophila | Lactobacillus rhamnosus |
| Hanseniaspora uvarum | Leuconostoc mesenteroides |
| Hanseniaspora valbyensis | Oenococcus oeni |
| Metschinikowia pulcherrima | Pediococcus ethanoliduran |
| Pichia guilliermondii | Pediococcus parvulus |
| Pichia membranafaciens | |
| Zygossaccharomyces bailii |
|
| Saccharomyces bayanus | |
| Saccharomyces cerevisiae | |
| Saccharomycodes ludwigi |
Por su parte, para la identificación de las bacterias lácticas se realiza en primer lugar una amplificación de un fragmento del ADN ribosómico 16S de aproximadamente 530 pares de bases (Werning y col. 2006). Posteriormente este fragmento se secuencia y la secuencia nucleotídica obtenida es alineada, empleando el programa informático BLAST y comparada con la base de datos NCBI (http://www.ncbi.nlm.nih.gov) para obtener la asignación de la bacteria a nivel de género o de especie (Figura 2). Hasta el momento se ha realizado la identificación de aproximadamente 400 bacterias lácticas que han sido clasificadas en nueve especies (Tabla 1).
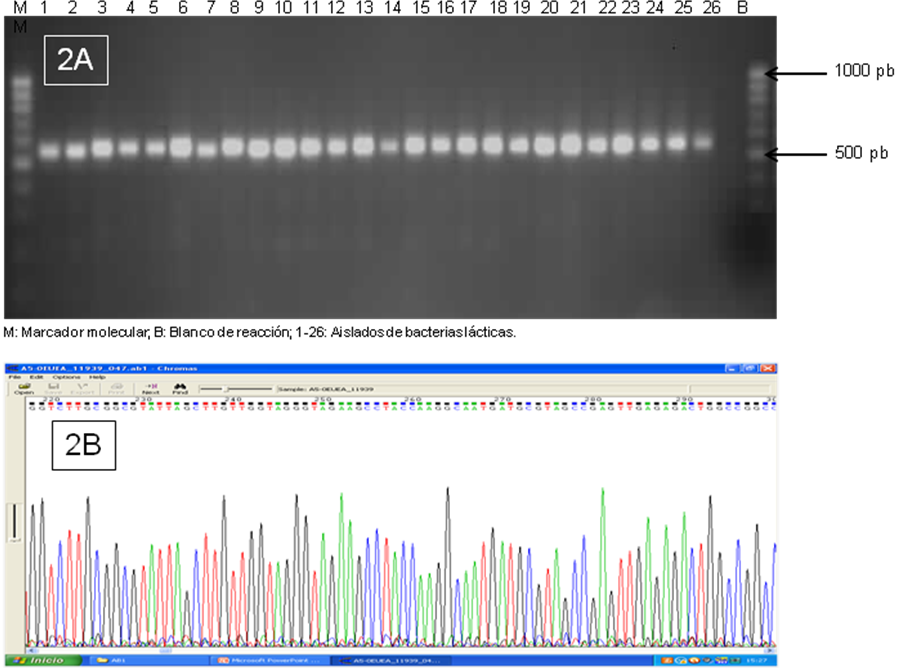
Figura 2: Perfiles de amplificación visualizados mediante electroforesis y electroferograma de la región ribosómica 16S de bacterias lacticas. 2A: Amplificados obtenidos en la reacción de PCR. 2B: Electroferograma derivado de la secuenciación del fragmento de ADN amplificado.
Caracterización molecular, fisiológica y tecnológica de recursos microbianos
Las actividades orientadas a caracterizar, es decir a diferenciar inequívocamente los distintos individuos o cepas de las Colecciones de Cultivo Microbianas y a determinar sus propiedades o atributos suelen abordarse por grupos microbianos. En este sentido, los recursos de origen sidrero se agrupan en levaduras Saccharomyces, levaduras no-Saccharomyces y bacterias lácticas.
La diferenciación de cepas se realiza mediante métodos moleculares (Figura 3). Así, en el caso de las levaduras Saccharomyces se utiliza el análisis de restricción del ADN mitocondrial (Querol y col., 1992). Dicha molécula posee un alto grado de variabilidad en las levaduras de este género y es muy estable durante los procesos de multiplicación. Las diferentes cepas se evidencian, mediante electroforesis, por la presencia de distintos perfiles de digestión. Para las levaduras no-Saccharomyces y bacterias lácticas, el polimorfismo entre individuos se evalúa por la presencia o ausencia de fragmentos de ADN amplificado cuando se utilizan cebadores o iniciadores de secuencia arbitraria y corta longitud (Zapparoli y col. 2000; Bujdosó y col. 2001).
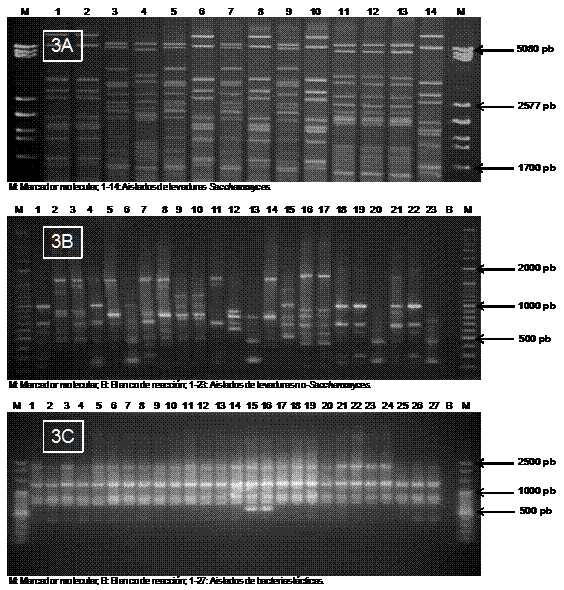
Figura 3: Caracterización molecular de recursos microbianos de origen sidrero. 3A: Perfiles de restricción del ADN mitocondrial de levaduras del género Saccharomyces. 3B: Perfiles de amplificación obtenidos mediante RAPD con el oligonucleótido RM13 de levaduras no-Saccharomyces. 3C: Perfiles de amplificación obtenidos mediante RAPD con el oligonucleótido M13 de bacterias lácticas.
La caracterización fisiológica y tecnológica de cepas genera información que puede ser utilizada para mejorar los procesos de elaboración y la calidad de los productos. En este sentido, para cada grupo microbiano se han analizado características acordes a las actividades o funciones que poseen durante la transformación del mosto de manzana (Figura 4).


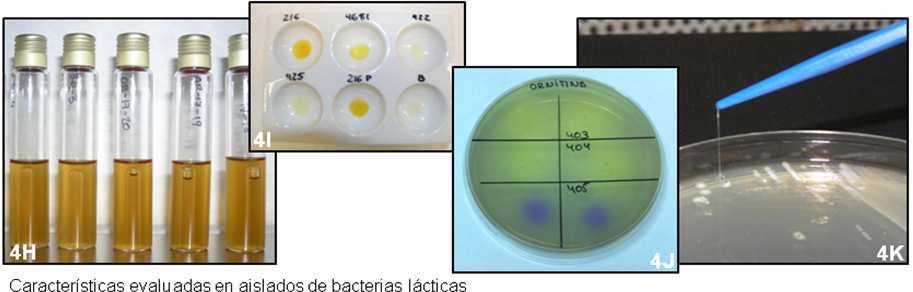
Figura 4. Caracterización fisiológica y tecnológica de recursos microbianos de origen sidrero. 4A: Poder fermentativo; 4B: Producción de ácido sulfhídrico; 4C: Características espumantes; 4D: Determinación del factor killer; 4E: Producción de aromas; 4F: Determinación actividad glicosidasa; 4G: Determinación actividad proteolítica; 4H: Carácter homo-heterofermentativo; 4I: Producción de precursores de carbamato de etilo; 4J: Producción de aminas biógenas; 4K: Determinación de fenotipo “ropy”.
Para las levaduras Saccharomyces, principales responsables de la fermentación de los azúcares del mosto, se han analizado características consideradas criterios discriminantes a la hora de realizar una selección de iniciadores o starters (Tabla 2). Así por ejemplo, en este género se ha evaluado la capacidad de las distintas cepas para producir y tolerar etanol, producir ésteres, alcoholes superiores y compuestos volátiles que influyen en el aroma de las sidras y otros aspectos como la producción de toxina killer o las características espumantes. En el caso de las levaduras no-Saccharomyces, productoras de bajas graduaciones alcohólicas y altas concentraciones de sustancias volátiles que influyen en el aroma de los fermentados, se ha analizado su tolerancia al etanol y la producción de actividades enzimáticas. Finalmente, para las bacterias lácticas, responsables de la transformación maloláctica y de importantes alteraciones en sidra (picado láctico, picado alílico y filado), las características determinadas han sido la producción de sustancias alterantes y nocivas para la salud, y la capacidad de metabolizar el ácido málico en cepas Oenococcus oeni y Lactobacillus plantarum/pentosus.
Tabla 2: Características fisiológicas y tecnológicas analizadas en recursos microbianos de origen sidrero.
| Levaduras Saccharomyces | Levaduras no-Saccharomyces | Bacterias lácticas |
| Tolerancia al etanol | Poder fermentativo | Carácter -heterofermentativo |
| Poder fermentativo | Producción de aromas | Producción de ácido acético |
| Producción de aromas | Determinación de actividad pectolítica | Producción de aminas biógenas |
| Producción de ácido acético | Determinación de actividad proteolítica | Producción de precursores de carbamato de etilo |
| Producción de dióxido de azufre | Determinación de actividad glicosidasa | Producción de polisacaridos extracelulares |
| Producción de ácido sulhídrico | Determinación del fenotipo ropy | |
| Determinación del factor killer | Rendimiento transformación maloláctica | |
| Capacidad de floculación | ||
| Caaracterísticas espumantes | ||
| Determinación de actividad glicosidasa |
Por último, cabe destacar que la información asociada a las acciones de investigación sobre la Colección de Cultivos Autóctonos del SERIDA ha propiciado el uso de recursos microbianos de origen sidrero en la mejora de procesos de elaboración de productos como la sidra natural, la sidra de segunda fermentación en botella o el aguardiente de magaya.
Agradecimientos
La conservación y caracterización de microorganismos de origen sidrero se ha realizado con ayuda de los proyectos RM2006-00008, RM2009-00005 y RTA2009-00111 financiados por el Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA) con fondos FEDER y PC04-24 financiado por la Fundación para el Fomento en Asturias de la Investigación Científica Aplicada y la Tecnología (FICYT).
Referencias bibliográficas
WERNIG, M.L.; IBARBURU, I.; DUEÑAS, M.T.; IRASTORZA, A.; NAVAS, J.; LOPÉZ, P. (2006). Pediococcus parvulus gtf Gene encoding the gtf glycosyltransferase and its application for specific PCR detection of ß-D-glucan-producing bacteria in foods and beverages. J. Food Protection. 69 (1): 161-169.
BUJDOSÓ, G.; CHRISTOPH, M; EGLI, M.; HENICK-KLING, T. (2001). Characterization of Hanseniaspora (Kloeckera) strains isolated in finger lakes wineries using physiological and molecular techniques. Food Technol. Biotechnol., 39: 83-91.
ESTEVE-ZARSOSO, B.; BELLOCH, C.; URUBURU, F.; QUEROL, A. (1999). Identification of yeast by RFLP analysis of the 5.8S rRNA gene and the two ribosomal internal transcribed spacers. Int. J. Syst. Bacteriol. 47: 341-344
QUEROL, A.; BARRIO, E.; HUERTA, T.; RAMON, D. (1992). Molecular monitoring of wine fermentations conducted by active dry yeast strains. Appl. Environ. Microbiol. 58 (9): 2948-2953.
ZAPPAROLI, G.; REGUANT, C.; BORDONS, A.; TORRIANI, S.; DELLAGLIO, F. (2000). Genomic DNA fingerprinting of Oenococcus oeni strains by pulsed-field gel electrophoresis and randomly amplified polymorphic DNA-PCR. Current Microbiol. 40: 351-355.
| Ficha Bibliográfica | |
|---|---|
| Título | Microorganismos de origen sidrero, recursos genéticos microbianos, al servicio de la biotecnología (II) |
| Autor/es | PANDO BEDRIÑANA, R.; VALDERAS HERRERO, M.T.; SUAREZ VALLES, B. |
| Año Publicación | 2014 |
| Área | |
| Revista/Serie | Tecnología Agroalimentaria. Boletín informativo del SERIDA |
| Referencia | N. 13. pp. 49-53 |
| Formato | |
| Depósito Legal | 2.617/1995 |
| ISSN | 1135-6030 |
| ISBN | |
| Ver/Descargar | |

